- Tabellen und Diagramme
- Günstige Netzarchitekturen für die Analyse
- Inhalt der CD-ROM
- Daten
- Extrahierte Zeitreihen
- Rohdaten Biologie
- Rohdaten DWD
- Rohdaten FH
- Sonstige Rohdaten
Tabelle .51: Netzarchitekturen und Analysefehler im Zeitraum vom 12.11.96 - 31.12.96, nach RMSE sortiert (Teil 1)
|
mape |
mre |
sres |
rmse |
theil |
Lern- funktion |
Skalierungs- funktion |
Hidden-Neuronen |
Topologie- modifikation |
|
1.8601 |
0.0186 |
3216.8000 |
8.4549 |
0.5593 |
BP_momentum |
linear |
10 |
vorS+shortC |
|
1.8611 |
0.0187 |
3243.5000 |
8.4899 |
0.5623 |
BPM_ln_cosh |
linear |
3 |
vorS+shortC |
|
1.8293 |
0.0183 |
3275.5000 |
8.5316 |
0.5656 |
BPM_ln_cosh |
linear |
5 |
vorS |
|
1.8290 |
0.0183 |
3301.2000 |
8.5650 |
0.5682 |
BP_momentum |
linear |
6 |
vorS+shortC |
|
1.8411 |
0.0184 |
3335.4000 |
8.6093 |
0.5704 |
BP_momentum |
linear |
7 |
vorS+shortC |
|
1.8926 |
0.0189 |
3341.0000 |
8.6165 |
0.5679 |
BPM_ln_cosh |
linear |
9 |
vorS |
|
1.8676 |
0.0187 |
3350.0000 |
8.6282 |
0.5719 |
BP_momentum |
linear |
9 |
none |
|
1.8529 |
0.0186 |
3359.5000 |
8.6404 |
0.5740 |
BP_momentum |
linear |
8 |
none |
|
1.8095 |
0.0182 |
3369.8000 |
8.6536 |
0.5745 |
BP_momentum |
linear |
7 |
vorS |
|
1.9499 |
0.0194 |
3371.1000 |
8.6552 |
0.5708 |
BP_momentum |
linear |
4 |
vorS+shortC |
|
1.7218 |
0.0173 |
3392.2000 |
8.6823 |
0.5766 |
BPM_ln_cosh |
linear |
4 |
none |
|
1.8764 |
0.0188 |
3422.6000 |
8.7212 |
0.5759 |
BPM_ln_cosh |
linear |
3 |
none |
|
1.7398 |
0.0175 |
3449.5000 |
8.7553 |
0.5820 |
Rprop |
linear |
7 |
vorS+shortC |
|
1.8693 |
0.0187 |
3457.1000 |
8.7650 |
0.5805 |
BP_momentum |
linear |
5 |
vorS+shortC |
|
1.7793 |
0.0178 |
3472.0000 |
8.7838 |
0.5825 |
BPM_ln_cosh |
linear |
7 |
none |
|
1.7793 |
0.0178 |
3472.0000 |
8.7838 |
0.5825 |
BPM_ln_cosh |
linear |
7 |
shortC |
|
1.8124 |
0.0183 |
3472.2000 |
8.7840 |
0.5834 |
BPM_ln_cosh |
linear |
7 |
vorS |
|
1.7680 |
0.0177 |
3490.7000 |
8.8074 |
0.5841 |
BPM_ln_cosh |
linear |
8 |
none |
|
1.7930 |
0.0180 |
3498.3000 |
8.8170 |
0.5849 |
BPM_ln_cosh |
linear |
6 |
none |
|
1.8708 |
0.0187 |
3504.4000 |
8.8248 |
0.5842 |
BP_momentum |
linear |
4 |
vorS |
|
1.8711 |
0.0187 |
3508.0000 |
8.8292 |
0.5855 |
BPM_ln_cosh |
linear |
3 |
vorS |
|
1.8780 |
0.0188 |
3561.2000 |
8.8959 |
0.5889 |
BPM_ln_cosh |
linear |
7 |
vorS+shortC |
|
1.8620 |
0.0187 |
3567.3000 |
8.9035 |
0.5908 |
BP_momentum |
linear |
3 |
vorS |
|
1.7502 |
0.0176 |
3579.7000 |
8.9190 |
0.5919 |
BPM_ln_cosh |
linear |
6 |
vorS |
|
1.9249 |
0.0192 |
3605.9000 |
8.9515 |
0.5928 |
BP_momentum |
linear |
6 |
vorS |
|
1.8637 |
0.0187 |
3637.4000 |
8.9906 |
0.5964 |
BPM_ln_cosh |
linear |
4 |
vorS |
|
1.8642 |
0.0188 |
3690.9000 |
9.0565 |
0.6012 |
BP_momentum |
linear |
8 |
vorS+shortC |
|
1.8447 |
0.0186 |
3749.2000 |
9.1278 |
0.6054 |
BPM_ln_cosh |
linear |
9 |
none |
|
1.8213 |
0.0184 |
3777.6000 |
9.1622 |
0.6092 |
BP_momentum |
linear |
3 |
none |
|
1.8038 |
0.0181 |
3786.6000 |
9.1731 |
0.6093 |
BP_momentum |
linear |
4 |
none |
|
2.0475 |
0.0203 |
3793.8000 |
9.1818 |
0.6039 |
BP_momentum |
linear |
5 |
vorS |
|
1.8249 |
0.0183 |
3844.6000 |
9.2432 |
0.6138 |
BP_momentum |
linear |
6 |
none |
|
1.9048 |
0.0192 |
3871.7000 |
9.2756 |
0.6169 |
BP_momentum |
linear |
5 |
none |
|
1.8140 |
0.0183 |
3874.0000 |
9.2784 |
0.6152 |
BPM_ln_cosh |
linear |
10 |
none |
|
1.7821 |
0.0180 |
3875.7000 |
9.2804 |
0.6170 |
Rprop |
linear |
7 |
none |
Tabelle .52: Netzarchitekturen und Analysefehler im Zeitraum vom 12.11.96 - 31.12.96, nach RMSE sortiert (Teil 2)
|
mape |
mre |
sres |
rmse |
theil |
Lern- funktion |
Skalierungs- funktion |
Hidden-Neuronen |
Topologie- modifikation |
|
1.7821 |
0.0180 |
3875.7000 |
9.2804 |
0.6170 |
Rprop |
linear |
7 |
shortC |
|
1.8918 |
0.0189 |
3883.7000 |
9.2901 |
0.6061 |
BP_momentum |
m -s -X |
7 |
none |
|
1.8918 |
0.0189 |
3883.7000 |
9.2901 |
0.6061 |
BP_momentum |
m -s -X |
7 |
shortC |
|
1.9777 |
0.0198 |
3884.9000 |
9.2914 |
0.6000 |
BPM_ln_cosh |
m -s |
4 |
none |
|
2.0640 |
0.0206 |
3884.9000 |
9.2915 |
0.6035 |
BP_momentum |
m -s |
6 |
vorS+shortC |
|
1.9200 |
0.0192 |
3893.2000 |
9.3013 |
0.6054 |
BPM_ln_cosh |
m -s -X |
7 |
none |
|
1.9200 |
0.0192 |
3893.2000 |
9.3013 |
0.6054 |
BPM_ln_cosh |
m -s -X |
7 |
shortC |
|
2.0509 |
0.0205 |
3920.8000 |
9.3343 |
0.6029 |
BP_momentum |
m -s |
7 |
vorS+shortC |
|
1.8291 |
0.0184 |
3938.5000 |
9.3553 |
0.6215 |
Rprop |
linear |
7 |
vorS |
|
1.8847 |
0.0190 |
3963.9000 |
9.3855 |
0.6198 |
BPM_ln_cosh |
linear |
6 |
vorS+shortC |
|
2.0179 |
0.0202 |
3967.0000 |
9.3891 |
0.6125 |
Rprop |
m -s -X |
7 |
vorS |
|
1.9249 |
0.0194 |
3973.1000 |
9.3963 |
0.6171 |
BPM_ln_cosh |
m -s -0 |
7 |
vorS+shortC |
|
2.0978 |
0.0209 |
3985.6000 |
9.4111 |
0.6224 |
BPM_ln_cosh |
linear |
10 |
vorS+shortC |
|
2.0159 |
0.0202 |
4007.0000 |
9.4363 |
0.6113 |
BPM_ln_cosh |
m -s |
10 |
none |
|
1.9019 |
0.0192 |
4044.6000 |
9.4805 |
0.6195 |
Rprop |
m -s -0 |
7 |
vorS |
|
2.0615 |
0.0207 |
4057.8000 |
9.4959 |
0.6138 |
BP_momentum |
m -s |
4 |
vorS+shortC |
|
2.0080 |
0.0202 |
4071.0000 |
9.5114 |
0.6117 |
BP_momentum |
m -s |
4 |
none |
|
1.9638 |
0.0197 |
4118.7000 |
9.5669 |
0.6186 |
BP_momentum |
m -s -X |
7 |
vorS+shortC |
|
1.9530 |
0.0198 |
4134.8000 |
9.5857 |
0.6313 |
Rprop |
m -s -0 |
7 |
vorS+shortC |
|
1.9896 |
0.0200 |
4136.3000 |
9.5874 |
0.6186 |
Rprop |
m -s |
7 |
vorS |
|
1.9426 |
0.0196 |
4155.3000 |
9.6094 |
0.6382 |
BPM_ln_cosh |
linear |
5 |
vorS+shortC |
|
2.0622 |
0.0207 |
4206.8000 |
9.6688 |
0.6313 |
BPM_ln_cosh |
m -s |
3 |
none |
|
1.9526 |
0.0197 |
4215.0000 |
9.6781 |
0.6423 |
BP_momentum |
linear |
8 |
vorS |
|
2.0782 |
0.0208 |
4228.6000 |
9.6937 |
0.6276 |
BPM_ln_cosh |
m -s |
9 |
none |
|
1.9170 |
0.0194 |
4240.9000 |
9.7079 |
0.6413 |
BPM_ln_cosh |
linear |
8 |
vorS+shortC |
|
2.0770 |
0.0209 |
4256.6000 |
9.7258 |
0.6279 |
BP_momentum |
m -s |
3 |
vorS+shortC |
|
2.0527 |
0.0206 |
4287.2000 |
9.7606 |
0.6336 |
BP_momentum |
m -s |
8 |
none |
|
2.0864 |
0.0210 |
4288.2000 |
9.7619 |
0.6292 |
BP_momentum |
m -s |
9 |
vorS+shortC |
|
1.9229 |
0.0195 |
4303.5000 |
9.7792 |
0.6439 |
BPM_ln_cosh |
linear |
4 |
vorS+shortC |
|
2.0659 |
0.0207 |
4314.6000 |
9.7918 |
0.6369 |
BPM_ln_cosh |
m -s |
7 |
none |
|
2.0659 |
0.0207 |
4314.6000 |
9.7918 |
0.6369 |
BPM_ln_cosh |
m -s |
7 |
shortC |
|
2.0813 |
0.0209 |
4357.7000 |
9.8406 |
0.6356 |
BP_momentum |
m -s |
10 |
none |
|
2.1253 |
0.0213 |
4358.7000 |
9.8417 |
0.6399 |
BP_momentum |
m -s |
7 |
none |
|
2.1253 |
0.0213 |
4358.7000 |
9.8417 |
0.6399 |
BP_momentum |
m -s |
7 |
shortC |
|
2.0546 |
0.0206 |
4359.9000 |
9.8431 |
0.6405 |
Rprop |
m -s -X |
7 |
none |
|
2.0546 |
0.0206 |
4374.5000 |
9.8595 |
0.6421 |
BPM_ln_cosh |
m -s |
8 |
none |
|
2.0895 |
0.0210 |
4374.6000 |
9.8597 |
0.6378 |
BPM_ln_cosh |
m -s |
5 |
none |
|
2.0682 |
0.0208 |
4379.6000 |
9.8653 |
0.6426 |
BP_momentum |
m -s |
3 |
none |
|
2.0770 |
0.0209 |
4384.8000 |
9.8711 |
0.6363 |
BP_momentum |
m -s |
9 |
none |
|
1.9243 |
0.0195 |
4385.9000 |
9.8724 |
0.6564 |
BPM_ln_cosh |
linear |
5 |
none |
|
2.0661 |
0.0207 |
4436.0000 |
9.9286 |
0.6384 |
BP_momentum |
m -s -X |
7 |
vorS |
|
2.1115 |
0.0212 |
4443.1000 |
9.9366 |
0.6412 |
BP_momentum |
m -s |
10 |
vorS+shortC |
|
1.9235 |
0.0195 |
4452.0000 |
9.9466 |
0.6538 |
BPM_ln_cosh |
m -s -0 |
7 |
vorS |
|
2.1046 |
0.0211 |
4473.6000 |
9.9706 |
0.6456 |
BPM_ln_cosh |
m -s |
6 |
none |
|
1.9599 |
0.0198 |
4495.1000 |
9.9945 |
0.6605 |
BPM_ln_cosh |
linear |
9 |
vorS+shortC |
|
2.0896 |
0.0210 |
4497.9000 |
9.9977 |
0.6459 |
Rprop |
m -s |
7 |
none |
|
2.0896 |
0.0210 |
4497.9000 |
9.9977 |
0.6459 |
Rprop |
m -s |
7 |
shortC |
|
1.9835 |
0.0200 |
4543.5000 |
10.0480 |
0.6631 |
Rprop |
m -s -0 |
7 |
none |
Tabelle .53: Netzarchitekturen und Analysefehler im Zeitraum vom 12.11.96 - 31.12.96, nach RMSE sortiert (Teil 2)
|
mape |
mre |
sres |
rmse |
theil |
Lern- funktion |
Skalierungs- funktion |
Hidden-Neuronen |
Topologie- modifikation |
|
1.9835 |
0.0200 |
4543.5000 |
10.0480 |
0.6631 |
Rprop |
m -s -0 |
7 |
shortC |
|
2.1302 |
0.0214 |
4701.2000 |
10.2210 |
0.6603 |
BP_momentum |
m -s |
5 |
vorS+shortC |
|
2.0533 |
0.0208 |
4701.1000 |
10.2210 |
0.6723 |
BPM_ln_cosh |
m -s -0 |
7 |
none |
|
2.0533 |
0.0208 |
4701.1000 |
10.2210 |
0.6723 |
BPM_ln_cosh |
m -s -0 |
7 |
shortC |
|
2.1553 |
0.0217 |
4745.8000 |
10.2700 |
0.6620 |
BP_momentum |
m -s |
6 |
none |
|
2.1456 |
0.0216 |
4772.1000 |
10.2980 |
0.6627 |
BP_momentum |
m -s |
5 |
none |
|
2.1522 |
0.0217 |
4837.6000 |
10.3680 |
0.6725 |
BP_momentum |
m -s |
8 |
vorS+shortC |
|
2.2541 |
0.0226 |
5116.0000 |
10.6620 |
0.6974 |
Rprop |
m -s |
7 |
vorS+shortC |
|
2.0994 |
0.0213 |
5163.5000 |
10.7120 |
0.7114 |
BP_momentum |
linear |
9 |
vorS |
|
2.4432 |
0.0241 |
5214.5000 |
10.7650 |
0.7041 |
BP_momentum |
linear |
9 |
vorS+shortC |
|
2.1879 |
0.0222 |
5428.5000 |
10.9830 |
0.7274 |
BP_momentum |
linear |
10 |
vorS |
|
2.1910 |
0.0223 |
5803.8000 |
11.3570 |
0.7444 |
BP_momentum |
m -s -0 |
7 |
vorS |
|
2.2928 |
0.0233 |
5827.8000 |
11.3800 |
0.7527 |
BP_momentum |
linear |
3 |
vorS+shortC |
|
2.5836 |
0.0254 |
5901.1000 |
11.4510 |
0.7481 |
BP_momentum |
linear |
10 |
none |
|
2.3007 |
0.0234 |
6008.6000 |
11.5550 |
0.7579 |
BP_momentum |
m -s -0 |
7 |
none |
|
2.3007 |
0.0234 |
6008.6000 |
11.5550 |
0.7579 |
BP_momentum |
m -s -0 |
7 |
shortC |
|
2.6109 |
0.0257 |
6157.0000 |
11.6970 |
0.7631 |
BP_momentum |
linear |
7 |
none |
|
2.6109 |
0.0257 |
6157.0000 |
11.6970 |
0.7631 |
BP_momentum |
linear |
7 |
shortC |
|
2.3144 |
0.0236 |
6408.0000 |
11.9330 |
0.7777 |
BP_momentum |
m -s -0 |
7 |
vorS+shortC |
|
2.5299 |
0.0252 |
6508.2000 |
12.0260 |
0.7888 |
Rprop |
m -s -X |
7 |
vorS+shortC |
|
2.7800 |
0.0273 |
6616.0000 |
12.1250 |
0.7930 |
BPM_ln_cosh |
linear |
8 |
vorS |
|
2.8117 |
0.0276 |
6662.5000 |
12.1680 |
0.7932 |
BPM_ln_cosh |
linear |
10 |
vorS |
|
4.3035 |
0.0442 |
17520.0000 |
19.7320 |
1.2769 |
BPM_ln_cosh |
m -s -X |
7 |
vorS+shortC |
|
4.3036 |
0.0442 |
17522.0000 |
19.7330 |
1.2769 |
BP_momentum |
m -s |
3 |
vorS |
|
4.3037 |
0.0442 |
17523.0000 |
19.7330 |
1.2769 |
BPM_ln_cosh |
m -s |
3 |
vorS+shortC |
|
4.3037 |
0.0442 |
17523.0000 |
19.7330 |
1.2769 |
BPM_ln_cosh |
m -s |
3 |
vorS |
|
4.3036 |
0.0442 |
17522.0000 |
19.7330 |
1.2769 |
BP_momentum |
m -s |
4 |
vorS |
|
4.3037 |
0.0442 |
17523.0000 |
19.7330 |
1.2769 |
BPM_ln_cosh |
m -s |
4 |
vorS+shortC |
|
4.3037 |
0.0442 |
17523.0000 |
19.7330 |
1.2769 |
BPM_ln_cosh |
m -s |
4 |
vorS |
|
4.3036 |
0.0442 |
17522.0000 |
19.7330 |
1.2769 |
BP_momentum |
m -s |
5 |
vorS |
|
4.3037 |
0.0442 |
17523.0000 |
19.7330 |
1.2769 |
BPM_ln_cosh |
m -s |
5 |
vorS+shortC |
|
4.3037 |
0.0442 |
17523.0000 |
19.7330 |
1.2769 |
BPM_ln_cosh |
m -s |
5 |
vorS |
|
4.3036 |
0.0442 |
17522.0000 |
19.7330 |
1.2769 |
BP_momentum |
m -s |
6 |
vorS |
|
4.3037 |
0.0442 |
17523.0000 |
19.7330 |
1.2769 |
BPM_ln_cosh |
m -s |
6 |
vorS+shortC |
|
4.3037 |
0.0442 |
17523.0000 |
19.7330 |
1.2769 |
BPM_ln_cosh |
m -s |
6 |
vorS |
|
4.3036 |
0.0442 |
17522.0000 |
19.7330 |
1.2769 |
BP_momentum |
m -s |
7 |
vorS |
|
4.3037 |
0.0442 |
17523.0000 |
19.7330 |
1.2769 |
BPM_ln_cosh |
m -s |
7 |
vorS+shortC |
|
4.3037 |
0.0442 |
17523.0000 |
19.7330 |
1.2769 |
BPM_ln_cosh |
m -s |
7 |
vorS |
|
4.3037 |
0.0442 |
17523.0000 |
19.7330 |
1.2769 |
BP_momentum |
m -s |
8 |
vorS |
|
4.3037 |
0.0442 |
17523.0000 |
19.7330 |
1.2769 |
BPM_ln_cosh |
m -s |
8 |
vorS+shortC |
|
4.3037 |
0.0442 |
17523.0000 |
19.7330 |
1.2769 |
BPM_ln_cosh |
m -s |
8 |
vorS |
|
4.3037 |
0.0442 |
17523.0000 |
19.7330 |
1.2769 |
BP_momentum |
m -s |
9 |
vorS |
|
4.3037 |
0.0442 |
17523.0000 |
19.7330 |
1.2769 |
BPM_ln_cosh |
m -s |
9 |
vorS+shortC |
|
4.3037 |
0.0442 |
17523.0000 |
19.7330 |
1.2769 |
BPM_ln_cosh |
m -s |
9 |
vorS |
|
4.3037 |
0.0442 |
17523.0000 |
19.7330 |
1.2769 |
BP_momentum |
m -s |
10 |
vorS |
|
4.3037 |
0.0442 |
17523.0000 |
19.7330 |
1.2769 |
BPM_ln_cosh |
m -s |
10 |
vorS+shortC |
|
4.3037 |
0.0442 |
17523.0000 |
19.7330 |
1.2769 |
BPM_ln_cosh |
m -s |
10 |
vorS |
|
4.3036 |
0.0442 |
17522.0000 |
19.7330 |
1.2769 |
BPM_ln_cosh |
m -s -X |
7 |
vorS |
Tabelle .54: Netzarchitekturen und Prognosefehler im Zeitraum vom 12.11.96 - 31.12.96, nach RMSE sortiert
|
mape |
mre |
sres |
rmse |
theil |
Lern- funktion |
Skalierungs- funktion |
Hidden-Neuronen |
Topologie- modifikation |
|
2.8852 |
0.0289 |
7648.8 |
13.3370 |
0.8673 |
BP_momentum |
my_sigma |
7 |
none |
|
2.8852 |
0.0289 |
7648.8 |
13.3370 |
0.8673 |
BP_momentum |
my_sigma |
7 |
shortC |
|
2.8415 |
0.0284 |
7766.7 |
13.4400 |
0.8740 |
BP_momentum |
my_sigma0 |
7 |
none |
|
2.8415 |
0.0284 |
7766.7 |
13.4400 |
0.8740 |
BP_momentum |
my_sigma0 |
7 |
shortC |
|
2.8849 |
0.0288 |
7786.5 |
13.4570 |
0.8751 |
BPM_ln_cosh |
my_sigma |
7 |
none |
|
2.8849 |
0.0288 |
7786.5 |
13.4570 |
0.8751 |
BPM_ln_cosh |
my_sigma |
7 |
shortC |
|
2.9170 |
0.0291 |
7906.3 |
13.5600 |
0.8818 |
Rprop |
my_sigma |
7 |
none |
|
2.9170 |
0.0291 |
7906.3 |
13.5600 |
0.8818 |
Rprop |
my_sigma |
7 |
shortC |
|
2.8250 |
0.0281 |
7939.9 |
13.5890 |
0.8836 |
BPM_ln_cosh |
linear |
7 |
vorS |
|
2.9369 |
0.0294 |
8014.3 |
13.6520 |
0.8878 |
Rprop |
my_sigma |
7 |
vorS |
|
2.8938 |
0.0289 |
8026.5 |
13.6620 |
0.8885 |
BPM_ln_cosh |
my_sigmaX |
7 |
none |
|
2.8938 |
0.0289 |
8026.5 |
13.6620 |
0.8885 |
BPM_ln_cosh |
my_sigmaX |
7 |
shortC |
|
2.9142 |
0.0292 |
8081.9 |
13.7100 |
0.8915 |
BP_momentum |
linear |
7 |
vorS |
|
2.9030 |
0.0292 |
8084.7 |
13.7120 |
0.8917 |
Rprop |
my_sigma0 |
7 |
vorS |
|
2.9065 |
0.0292 |
8217.4 |
13.8240 |
0.8990 |
Rprop |
my_sigma0 |
7 |
vorS+shortC |
|
2.9093 |
0.0291 |
8282.5 |
13.8790 |
0.9025 |
BP_momentum |
linear |
7 |
vorS+shortC |
|
2.9237 |
0.0291 |
8371.6 |
13.9530 |
0.9074 |
BP_momentum |
my_sigmaX |
7 |
none |
|
2.9237 |
0.0291 |
8371.6 |
13.9530 |
0.9074 |
BP_momentum |
my_sigmaX |
7 |
shortC |
|
3.0075 |
0.0302 |
8375.2 |
13.9560 |
0.9075 |
Rprop |
my_sigma0 |
7 |
none |
|
3.0075 |
0.0302 |
8375.2 |
13.9560 |
0.9075 |
Rprop |
my_sigma0 |
7 |
shortC |
|
3.0053 |
0.0302 |
8417.0 |
13.9910 |
0.9098 |
BPM_ln_cosh |
linear |
7 |
vorS+shortC |
|
2.9677 |
0.0296 |
8453.0 |
14.0210 |
0.9118 |
Rprop |
my_sigmaX |
7 |
vorS |
|
3.0006 |
0.0301 |
8472.5 |
14.0370 |
0.9128 |
BPM_ln_cosh |
my_sigma0 |
7 |
none |
|
3.0006 |
0.0301 |
8472.5 |
14.0370 |
0.9128 |
BPM_ln_cosh |
my_sigma0 |
7 |
shortC |
|
2.9992 |
0.0301 |
8536.4 |
14.0900 |
0.9162 |
BPM_ln_cosh |
my_sigma0 |
7 |
vorS+shortC |
|
3.0587 |
0.0306 |
8540.7 |
14.0930 |
0.9165 |
Rprop |
my_sigma |
7 |
vorS+shortC |
|
2.9874 |
0.0299 |
8835.7 |
14.3350 |
0.9322 |
Rprop |
linear |
7 |
vorS+shortC |
|
3.0680 |
0.0309 |
8847.5 |
14.3440 |
0.9328 |
BPM_ln_cosh |
my_sigma0 |
7 |
vorS |
|
3.0058 |
0.0299 |
8866.1 |
14.3590 |
0.9338 |
Rprop |
my_sigmaX |
7 |
none |
|
3.0058 |
0.0299 |
8866.1 |
14.3590 |
0.9338 |
Rprop |
my_sigmaX |
7 |
shortC |
|
3.0171 |
0.0303 |
8872.1 |
14.3640 |
0.9341 |
BP_momentum |
my_sigma0 |
7 |
vorS+shortC |
|
3.1153 |
0.0315 |
9110.9 |
14.5560 |
0.9466 |
BP_momentum |
my_sigma0 |
7 |
vorS |
|
3.0607 |
0.0307 |
9325.8 |
14.7270 |
0.9577 |
Rprop |
linear |
7 |
vorS |
|
3.1720 |
0.0320 |
9436.1 |
14.8140 |
0.9633 |
BPM_ln_cosh |
linear |
7 |
none |
|
3.1720 |
0.0320 |
9436.1 |
14.8140 |
0.9633 |
BPM_ln_cosh |
linear |
7 |
shortC |
|
3.1092 |
0.0311 |
9553.1 |
14.9050 |
0.9693 |
Rprop |
linear |
7 |
none |
|
3.1092 |
0.0311 |
9553.1 |
14.9050 |
0.9693 |
Rprop |
linear |
7 |
shortC |
|
3.1368 |
0.0314 |
9669.5 |
14.9960 |
0.9752 |
BP_momentum |
linear |
7 |
none |
|
3.1368 |
0.0314 |
9669.5 |
14.9960 |
0.9752 |
BP_momentum |
linear |
7 |
shortC |
|
3.2304 |
0.0321 |
10169.0 |
15.3780 |
1.0000 |
Rprop |
my_sigmaX |
7 |
vorS+shortC |
|
3.3333 |
0.0332 |
10862.0 |
15.8930 |
1.0335 |
BP_momentum |
my_sigmaX |
7 |
vorS+shortC |
|
3.3602 |
0.0333 |
11495.0 |
16.3500 |
1.0632 |
BP_momentum |
my_sigmaX |
7 |
vorS |
|
4.2565 |
0.0437 |
16560.0 |
19.6250 |
1.2762 |
BPM_ln_cosh |
my_sigmaX |
7 |
vorS |
|
4.2572 |
0.0437 |
16565.0 |
19.6270 |
1.2763 |
BPM_ln_cosh |
my_sigmaX |
7 |
vorS+shortC |
|
4.2589 |
0.0437 |
16579.0 |
19.6360 |
1.2769 |
BP_momentum |
my_sigma |
7 |
vorS+shortC |
Aufgrund der Fülle an anfallenden Daten und Ergebnissen war es nicht möglich, sämtliche Zeitreihen, Tabellen, Diagramme und Skripte in schriftlicher Form mit in diese Arbeit aufzunehmen. Um diese dennoch aufzuführen, wurden sie auf eine CD-ROM gebrannt, die dieser Arbeit beiliegt.
Übersicht über die Verzeichnisstruktur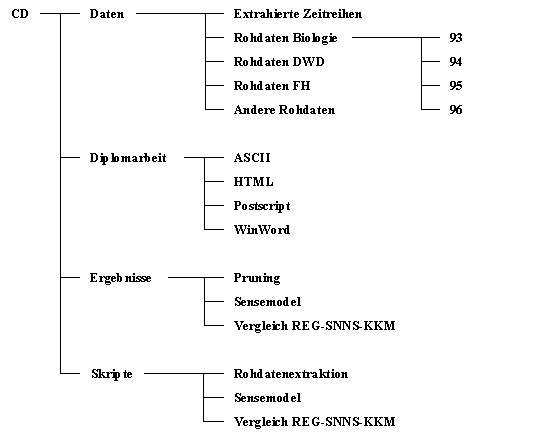
Abbildung .13: Übersicht über die Verzeichnisstruktur der CD-ROM
Das Datenverzeichnis gliedert sich in Rohdaten (verschiedene Quellen) und extrahierte Zeitreihen, die im Rahmen der Arbeit Verwendung fanden.
Enthält die verwendeten Datenreihen. Alle Datenreihen liegen im ASCII-Format vor und enthalten 1827 Zeilen, entsprechend dem betrachteten Zeitraum. Fehlwerte werden analog zu SAS als Punkt dargestellt. Der Name der Zeitreihendateien setzt sich wie folgt zusammen:
AAA-BBB-9296.txt
Dabei gibt AAA das Kürzel der Zeitreihe an (zwei oder mehr Buchstaben), BBB steht für die Datenquelle (DWD, FH, BIO oder ART für artifizielle selbsterstellte Zeitreihen).
Enthält für die Jahre 1993-1996 die Tagesdateien der CO2-Werte vom Fachbereich Biologie in entsprechenden Unterverzeichnissen. Die Werte für 1992 standen nicht unmittelbar zur Verfügung, sie wurden der Arbeit von Tillmanns entnommen [Til 93] und finden sich im Verzeichnis "Sonstige Rohdaten" (Siehe 7.3.2.5)in der Datei
daymean.92.Hier finden sich die Daten des Deutschen Wetterdienstes. Für die Jahre 1992 und 1993, sowie für den Zeitraum von 1994-1996 existieren ASCII-Dateien in verschiedenen Formaten. Sie heißen
sy92.out, sy93.out und sy94x96.dat. Die Daten liegen stundenweise vor.Die Wetterdaten von der Fachhochschule liegen als selbsterklärende Excel-Datei (Version 97) in der Datei
Wetterdaten FH.xls als Tageswerte vor.Enthält die Dateien
daymean.92 und daymean.93 aus der Arbeit von Tillmanns [Til 93], die u.a. die CO2-Werte von 1992 und 1993 als Tageswerte enthalten.Hier findet sich der Text der Diplomarbeit, in vier verschiedenen Formaten: ASCII, HTML, Postscript Color und WinWord-Format (Version 97). Das Titelblatt befindet sich jeweils in einer gesonderten Datei mit Namen
Titelblatt.* (* = txt, doc, etc.).diplomarbeit.txt
: Die Arbeit in ASCII (MS-DOS) ohne Abbildungen und Tabellen.diplomarbeit.htm
: Die Arbeit in HTML mit Abbildungen und Tabellen. Diese Datei ist das Zentraldokument. Die Konvertierung erfolgte mit WinWord 97. Andere Dateien sind Filialdokumente und Abbildungen.diplomarbeit.ps
: Die Arbeit in (farbigem) Postscript.